大鼠睫状神经营养因子siRNA的设计及其有效性验证的体外实验方法
热度0票 浏览102次
时间:2011年3月24日 13:35
【关键词】 RNA干扰; 睫状神经营养因子(CNTF); 大鼠; 体外实验
RNA干扰(RNAi)是近年发展起来的一项具有巨大医疗应用潜力的反向遗传学技术[1]。该技术有双链干扰RNA设计与合成、序列有效性的体外实验验证和体内实验等重要环节[2]。其中,前两个环节是保证实验成功的先决条件。与植物和低等动物不同,在哺乳动物细胞内进行RNA干扰的条件更加苛刻,尚存在许多困难。比如,体外实验中,小干扰 RNA(siRNA)的设计和有效性验证必须依据特定的规律[3-5],并且对操作的要求较高。尽管如此,我们在实验中不断总结经验教训,认为很多siRNA的设计、合成和效应验证在操作上有很强的规律可循,具备一定经验后就能事半功倍。因此,我们以大鼠睫状神经营养因子 (ciliary neurotrophic factor, CNTF)的RNA干扰为例,介绍siRNA设计合成及其体外有效性验证的全过程,,希望这些经验能够为其他实验人员提供一些有帮助的信息。
发表论文网
1 材料和方法
1.1 大鼠CNTF siRNA分子的设计合成和修饰
在PubMed主页(网址http:∥www.ncbi.nlm.nih.gov/pubmed/)中,大鼠CNTF的cDNA序列链接 Accession号为 “NM_013166”。进入Invitrogen公司的BLOCK?iTTM RNAi Designer主页,选择“siRNA”设计模式(即19nt靶序列选择),然后依次进行如下操作:将大鼠CNTF mRNA的Accession号输入到“Accession number”对话框中,勾选“Open reading frame(ORF)”选项(表示在开放读码框中寻找靶序列),选择物种为“Rat?Rattus norvegicus”,选定G/C百分比为35%~55%[3],选定“Default motif pattern”,最后点击设计按钮。系统自动给出多个候选靶序列,并根据每个序列符合规则的程度给出各自评分。本实验选择评分较高的3个靶cDNA序列依次排列,序列1:GCAAACACCTCTGACCCTT;序列2:GGTGACTTCCATCAGGCAA;序列 3:TCCATGACCTTCGTGTCAT。通过网页上的“siRNA to Stealth RNAiTM siRNA”选项将三者转换为25nt序列(向5′?端延长6nt)后,序列1:CGCAGAGCAAACACCTCTGACCCTT,序列 2:ACTGAAGGTGACTTCCATCAGGCAA,序列3:GGTCTATCCATGACCTTCGTGTCAT;系统同时自动设计出对照序列。根据靶序列设计出3个siRNA分子。siRNA1正义链:5′?CGCAGAGCAAACACCUCUGACCCUU?3′,反义链:3′ ?GCGUCUCGUUUGUGGAGACUGGGAA?5′;siRNA2正义链:5′?ACUGAAGGUGACUUCCAUCAGGCAA?3′,反义链:3′?UGACUUCCACUGAAGGUAGUCCGUU?5′;siRNA3正义链:5′? GGUCUAUCCAUGACCUUCGUGUCAU?3′,反义链:3′?CCAGAUAGGUACUGGAAGCACAGUA?5′。转染操作的对照序列(按cDNA)为:CGCACGAACAACTCCCAGTCGACTT,同上设计出对照siRNA分子。本实验的siRNA均为25nt两端齐平设计。3个siRNA分子及其对照物均由Invitrogen公司化学合成,脱盐法提纯,5′?端行Cy3荧光基团标记。
1.2 其他主要试剂用品
Lipofectamine 2000(Invitrogen),DMEM培养液(Hyclone),兔抗大鼠GFAP抗体(Sigma),兔抗大鼠Vimentin抗体(博奥森),兔抗大鼠S100抗体(博奥森),兔抗大鼠CNTF抗体(博奥森),胰酶(Sigma),Cy3标记羊抗兔第二抗体(中杉金桥),反转录和PCR试剂盒 (Fermenta);DNA标准参照物(华美),24孔培养板(Costar)。
1.3 主要仪器
高压蒸汽灭菌器(Sanyo),倒置荧光显微镜(Zeiss),数码相机(Olympus,带专用订制接口)超净工作台(苏州净化),二氧化碳孵箱(Sanyo),PCR仪(Bio?Rad),水平电泳仪(北京六一),微量加样枪(DISHI),凝胶成像系统(ChemiDoc),低温冰箱 (Sanyo),离心机(Bio?Rad)。
1.4 实验动物发表论文网
成年健康SD大鼠,由四川大学实验动物中心提供。雌雄合笼使其自然交配。雌鼠孕期给予孕鼠全价饲料,饮水经煮沸消毒处理并可自由摄取,饲养间环境温度18~24 ℃,自然光照。分娩后3天(本实验3种细胞的培养均取自该日龄),取乳鼠行断颈处死,整体浸泡于70%乙醇30 min后,置于超净台中的无菌培养皿内,解剖取材进行细胞培养。
1.5 细胞培养
本实验分别培养了施万细胞、嗅鞘细胞和成纤维细胞。施万细胞取自坐骨神经干,操作程序参考Manson 等[6]的方法;待细胞形态完全恢复梭形(大约3 d)即进行传代,16 h后单独用Lipofectamine 2000模拟转染细胞(见“1.6”),继续培养72 h,用S100免疫组化鉴定并观察。嗅鞘细胞取自嗅球,采用李绍波等的操作程序[7],纯化后培养7 d进行传代,16 h后同上用Lipofectamine 2000处理,继续培养72 h,用GFAP与p75鉴定并观察。成纤维细胞取自肾脏,参考Alvarez等的方法[8],血清浓度5%,细胞融合率达90%时进行传代,传代后处理同上,用Vimentin免疫组化鉴定并观察。以上细胞在纯化培养完成后,消化转瓶的过程中不减少细胞总数;所有培养液体不加抗生素。细胞观察计数在倒置显微镜下拍照并用ImagePro 5.0软件辅助分析,记录每高倍(40×10)视野下细胞的面积占视野总面积的比例,作为细胞的融合度(conflurency);随机计数5个视野(视野不能含有培养板的底部边缘)后取平均值作为该孔的融合度;一共计数6个孔取平均作为本组的细胞融合度。
1.6 siRNA体外转染操作
成纤维细胞以3×105个/ml的密度传代一次后,待细胞融合率达50%时,常规胰酶消化,不分瓶直接重新接种于D?多聚赖氨酸铺底的24孔板,每孔加入不含血清的DMEM培养基0.2 ml。其中6个孔在传代后16 h时进入转染程序,另6个在传代后48 h时进行。转染时,所有液体、容器和有可能接触标本的用具都要用DEPC处理后行高压灭菌。首先,摇匀Lipofectamine 2000原液,取6 μl,用300 μl DMEM培养基稀释,摇匀后室温(20 ~ 25 ℃)孵育5 min。然后取20 μmol/L siRNA原液6 μl,用300 μl DMEM培养基稀释于Eppendorf管中,轻轻翻转混匀。将Lipofectamine 2000稀释液和siRNA稀释液混合,轻轻翻转混匀,室温下孵育20 min。在此期间如果液体变浑浊并不影响效果。弃去培养板孔中的培养基,立即加入上述混合液,每孔100 μl。对照siRNA的转染操作方法与上述相同。转染过程需要避强光。转染后,细胞置于原培养条件(37 ℃,5% CO2)下继续培养,6 h后在倒置荧光显微镜下观察是否成功转染;分24,48和72 h三个时间点取材检测,每个时间点检测12个孔,其中6个为待检siRNA孔,6个为对照孔。
1.7 RT?PCR分析
用Premier 5.0软件设计大鼠β?肌动蛋白和CNTF的引物序列。β?肌动蛋白引物上游序列:5′?GTAAAGACCTCTATGCCAACA?3′,下游序列:5′?GGACTCATCGTACTCCTGCT?3′,扩增产物预计227 bp;CNTF引物上游序列:5′?CTTTCGCAGAGCAAACACCT?3′,下游序列:5′?CATCCCATCAGCCTCATTTT?3′ ,扩增产物预计422 bp。按说明书指示的流程进行操作,反应体系的总体积为25 μl;PCR扩增条件:94℃预变性(3 min),以94℃变性45 s,51℃退火45 s,72℃延伸1 min为一个循环,35个循环后,72℃延伸5 min。15 g/L琼脂糖凝胶电泳分离扩增产物,在凝胶成像仪上观察拍照,用ImagePro 5.0软件测定并计算CNTF与β?肌动蛋白扩增产物条带的平均灰度与条带面积乘积的比值,作为CNTF mRNA的相对含量。发表论文网
1.8 免疫荧光组织化学检测
将细胞培养板内的液体倒掉,用D?Hanks液快速漂洗2次,加入4℃预冷的95%乙醇铺满培养板底面,固定15 min。倾去乙醇,用PBS漂洗3次,每次2 min。滴加2%牛血清白蛋白PBST溶液封闭15 min,倾去后不洗直接滴加用PBS 1∶200稀释的CNTF抗体工作液,37℃孵育1 h后转入4℃冰箱孵育16 h。PBS漂洗3次,每次5 min。滴加PBS 1∶200稀释的荧光二抗,37℃避光孵育1 h。PBS避光漂洗3次,每次3 min。倒置荧光显微镜下观察、测定细胞的平均荧光强度(灰度)值并照相。
1.9 数据统计分析
用SPSS 16.0软件进行数据分析,采用组间t检验,P<0.05认为差别具有统计学意义。数据导入Excel 2003绘制直方图。
2 结 果
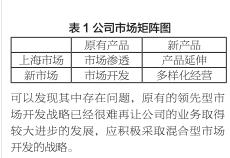
2.1 经过传代和Lipofectamine 2000处理后成纤维细胞的融合度最高,符合转染要求
经过传代和Lipofectamine 2000处理,如图1所示,施万细胞的融合度大大下降,嗅鞘细胞的融合度也明显降低,两者都不能满足常规siRNA转染的要求。成纤维细胞经过两次传代,细胞密度增加,细胞生长良好,满足转染要求。
2.2 在传代后16 h时进行转染操作,siRNA能够成功进入成纤维细胞
转染操作6 h后观察,如图2,传代后16 h转染组成纤维细胞90%以上带有红色荧光标记,证实siRNA已经成功转染成纤维细胞。传代后48 h转染组成纤维细胞很少见到荧光标记。
2.3 不同siRNA分子和不同转染条件下CNTF免疫荧光强度的变化
如图3所示,在传代后16 h进行转染操作,转染后24,48,72 h这3个时间点,3种核苷酸序列不同的siRNA分子所致的CNTF免疫荧光强度降低的程度是不同的。其中,siRNA2组各时间点CNTF免疫荧光强度无显著改变;siRNA1和siRNA3组可见在72 h时CNTF免疫荧光强度下降显著(P<0.01),两组分别下降至对照siRNA转染组的82%和61%。
2.4 siRNA3转染后CNTF mRNA水平显著下降
如图4,5,经RT?PCR分析可见,siRNA3转染后24 h,成纤维细胞的CNTF mRNA已经显著降低到对照siRNA转染组的14%,此后一直维持低水平。
3 讨 论发表论文网
本实验的主要内容为RNA干扰技术中siRNA的设计和有效性检验环节的操作。通过一系列操作,成功筛选出针对大鼠CNTF的相对有效的 siRNA3分子,并在最佳条件下使体外培养的成纤维细胞的CNTF mRNA和蛋白水平分别降低到对照siRNA转染组的14%和61%。
在包括CNTF等多种分子在内的RNAi实验中,我们根据操作经验,总结出大致相似的如下几个步骤。首先,根据目标蛋白的mRNA全长序列,按照经验规则[3-5]选择候选靶序列及其对照序列,并完成同源性检测;靶序列一般选择 3个,如果后续试验发现全部无效再作补充,否则数量多了会大大增加开支。选择靶序列和设计siRNA有很多自动化工具可用,我们应用后认为 Invitrogen公司的BLOCK?iTTM RNAi Designer比较全面和方便。此外,像麻省理工学院Whitehead研究所的siRNA设计程序和日本基因网SiDirect siRNA序列分析程序等得出的结果也值得参考,以上程序都可以在互联网上免费使用。siRNA分子可以是传统的19nt加上?dTdT等突出尾巴,也可以不采用19nt突出结构,评判是否有效的唯一方法就是通过实验验证。第二,根据靶序列设计出siRNA分子,确定siRNA分子的修饰类型,并交由专门的公司或实验室合成;siRNA合成修饰不是本文的重点内容,大多数研究形态或者功能的实验室也不需要常规进行这些操作。第三,通过文献查阅确定哪些细胞表达目标蛋白,然后用体外培养预实验确定:这些细胞中哪种或哪些细胞既能表达目标蛋白又比较容易转染。需要注意的是,为了检测细胞的生长特性和存活能力,传代过程不要降低细胞的总数。比如本实验通过体外培养发现,成纤维细胞高表达CNTF,而且传代后经过Lipofectamine 2000作用,仍然能生长和增殖,最符合转染操作的要求。如果有现成的细胞株或细胞系可以利用,仍然有必要进行上述预实验,因为细胞系或细胞株在多次传代后有可能丢失某些分子特点,甚至于其来源和属性都可能存在疑问[9]。如果实验室已经有相应细胞的培养经验,这一步上将会节约大量时间。第四,细胞消化传代(植板时不要减少细胞密度)并于16 h左右进行转染操作,大约6 h后镜下观察。之所以要在消化后16 h左右进行转染,是因为经过实验我们发现,细胞生长时间过长或过短,常规脂质体转染的成功率会大大下降。转染后6 h首次观察的目的是借助siRNA分子修饰基团(如荧光基团或酶)的成像确定siRNA是否已经进入细胞内;成功转染的细胞数应该在细胞总数的70%以上才具有意义[10]。至于siRNA的有效浓度,我们认为有必要进行预实验。因为浓度太低没有明确的干扰效果,浓度太高会因Lipofectamine 2000用量的增加而致使较多的细胞死亡。本文采用的浓度就是CNTF体外干扰的最佳浓度(本文中未显示该项预实验的数据)。第五,在转染成功的前提下继续培养,分多个时间点对各组细胞进行目标蛋白的免疫荧光组织化学检测,选出干扰效果最好的siRNA分子。时间点的选择可从转染后24 h开始,到72 h结束,只要有效,一般都能看到荧光强度减弱。虽然在哺乳动物细胞,常规脂质体转染的RNAi由于siRNA不能在细胞内复制和长期存在[11],其维持时间往往不超过170 h,但在siRNA分子有效性检测的环节,时间点没有必要选择那么长。最后,设计目标蛋白对应mRNA的RT?PCR引物(如果能从文献上或本实验室的数据库中获得准确信息更好),用通过免疫荧光检测选出的最有效的siRNA进行体外RNAi操作,分时间点取出细胞进行RT?PCR检测,进一步验证 mRNA水平是否被显著降低。
我们在siRNA有效性验证实验中,并没有先检测mRNA再检测蛋白,而是先通过免疫荧光检测找出最能降低蛋白表达水平的siRNA分子。这样做的原因有两方面:其一,蛋白水平的变化直接与功能相关,后续实验中RNAi是否有意义和能否起到人工干预效果,取决于蛋白表达是否降低,而不只是 mRNA水平的下降;其二,RT?PCR不论从成本还是操作的繁琐程度来说,都要高于免疫荧光检测。因此,我们在有效性验证的常规实验中反其道而行之,先看蛋白变化并找出最有效的siRNA分子,再看这种变化是否有mRNA水平降低这个基础条件,从而最终肯定所选siRNA分子的干扰效能。这样既快捷又节省费用。如果看不到免疫荧光强度的明显改变,再做一下蛋白质印迹是值得的。某些分子的RNAi操作中,mRNA水平降低后,蛋白水平始终不下降。虽然有的体内实验尝试每隔24 h到48 h反复转染取得了效果[12],但是我们在实践中发现这一做法在体外实验中经常是不能见效的,Lipofectamine 2000可能有一定细胞毒性,多次转染后细胞数量有明显下降(本文未显示该数据),况且这样达到的干扰效果即使用到体内实验中也将没有实际用途。所以如果发现这类蛋白水平难以降低的目标蛋白分子,往往有必要考虑改变转染方法,甚至调整实验的技术路线。
siRNA的脂质体转染技术虽然比较“传统”,但目前是进行体外RNAi操作的最常规且相当简便的方法。除了用具与液体必须杜绝RNAse外并没有需要特别小心的问题。成功地验证一个功能性的siRNA分子,就意味着在某个目标蛋白的研究中多了一个工具,并且根据相应的靶mRNA序列还能够设计出同样有效的短发夹状RNA(shRNA)分子,通过病毒载体等其他手段进行RNAi实验。因此掌握了siRNA的设计和有效性验证方法,就为RNAi实验奠定了基础。但是,本文的方法并不适用于所有的分子。这套方法对于存在以下问题的分子不适合:①只在神经细胞或其他某些难以分离传代的细胞上表达,这种情况需要使用病毒载体进行转染;②mRNA序列不长缺少选择余地,或者选择的靶序列经实验验证无一有效,或者设计的siRNA与管家基因有冲突,这些情况解决起来尚有困难;③目标蛋白半衰期太长,或者细胞内有大量储备,一时难以消耗掉,这种情况下有可能观察不到免疫荧光强度的降低,如有必要需直接用 RT?PCR检测来筛选有功能的siRNA分子。
发表论文网
【参考文献】
[1] Dykxhoorn DM, Palliser D, Lieberman J. The silent treatment: siRNAs as small molecule drugs[J]. Gene Ther, 2006,13(6): 541-552.
[2] Behlke MA. Progress towards in vivo use of siRNAs[J]. Mol Ther, 2006, 34(4): 644-670.
[3] Aronin N. Target selectivity in mRNA silencing[J]. Gene Ther, 2006,13(6): 509-516.
[4] Schwarz DS, Hutvágner G, Du T, et al. Asymmetry in the assembly of the RNAi enzyme complex[J]. Cell, 2003, 115(2):199-208.
[5] Pei Y, Tuschl T. On the art of identifying effective and specific siRNAs[J]. Nature Methods, 2006, 3(9): 670-676.
[6] Mason PW, Atlema BL, De Vries GH. Isolation and characterization of neonatal Schwann cells from cryopreserved rat sciatic nerves[J]. J Neurosci Res, 1992, 31(4): 731-744.
[7] 李绍波, 章 为, 周 雪,等. 成年大鼠嗅鞘细胞的原代培养与鉴定[J]. 四川大学学报: 医学版, 2007, 38(5): 717-719.
[8] Alvarez RJ, Sun MJ, Haverty TP, et al. Biosynthetic and proliferative characteristics of tubulointerstitial fibroblasts probed with paracrine cytokines[J]. Kidney Int, 1992, 41(1): 14-23.
[9] 弗雷谢尼. 动物细胞培养: 基本技术指南[M]. 北京: 科学出版社, 2004: 438.
[10] Tuschl T, Zamore PD, Lehmann R, et al. Targeted mRNA degradation by double?stranded RNA in vitro[J]. Genes Dev, 1999, 13(24): 3191-3197.
[11] Zamore PD. RNA interference: listening to the sound of silence[J]. Nat Struct Biol, 2001, 8(9): 746-750.
发表论文网
[12] Dorn G, Patel S, Wotherspoon G, et al. siRNA relieves chronic neuropathic pain[J]. Nucleic Acids Research, 2004, 32(5): e49.
RNA干扰(RNAi)是近年发展起来的一项具有巨大医疗应用潜力的反向遗传学技术[1]。该技术有双链干扰RNA设计与合成、序列有效性的体外实验验证和体内实验等重要环节[2]。其中,前两个环节是保证实验成功的先决条件。与植物和低等动物不同,在哺乳动物细胞内进行RNA干扰的条件更加苛刻,尚存在许多困难。比如,体外实验中,小干扰 RNA(siRNA)的设计和有效性验证必须依据特定的规律[3-5],并且对操作的要求较高。尽管如此,我们在实验中不断总结经验教训,认为很多siRNA的设计、合成和效应验证在操作上有很强的规律可循,具备一定经验后就能事半功倍。因此,我们以大鼠睫状神经营养因子 (ciliary neurotrophic factor, CNTF)的RNA干扰为例,介绍siRNA设计合成及其体外有效性验证的全过程,,希望这些经验能够为其他实验人员提供一些有帮助的信息。
发表论文网
1 材料和方法
1.1 大鼠CNTF siRNA分子的设计合成和修饰
在PubMed主页(网址http:∥www.ncbi.nlm.nih.gov/pubmed/)中,大鼠CNTF的cDNA序列链接 Accession号为 “NM_013166”。进入Invitrogen公司的BLOCK?iTTM RNAi Designer主页,选择“siRNA”设计模式(即19nt靶序列选择),然后依次进行如下操作:将大鼠CNTF mRNA的Accession号输入到“Accession number”对话框中,勾选“Open reading frame(ORF)”选项(表示在开放读码框中寻找靶序列),选择物种为“Rat?Rattus norvegicus”,选定G/C百分比为35%~55%[3],选定“Default motif pattern”,最后点击设计按钮。系统自动给出多个候选靶序列,并根据每个序列符合规则的程度给出各自评分。本实验选择评分较高的3个靶cDNA序列依次排列,序列1:GCAAACACCTCTGACCCTT;序列2:GGTGACTTCCATCAGGCAA;序列 3:TCCATGACCTTCGTGTCAT。通过网页上的“siRNA to Stealth RNAiTM siRNA”选项将三者转换为25nt序列(向5′?端延长6nt)后,序列1:CGCAGAGCAAACACCTCTGACCCTT,序列 2:ACTGAAGGTGACTTCCATCAGGCAA,序列3:GGTCTATCCATGACCTTCGTGTCAT;系统同时自动设计出对照序列。根据靶序列设计出3个siRNA分子。siRNA1正义链:5′?CGCAGAGCAAACACCUCUGACCCUU?3′,反义链:3′ ?GCGUCUCGUUUGUGGAGACUGGGAA?5′;siRNA2正义链:5′?ACUGAAGGUGACUUCCAUCAGGCAA?3′,反义链:3′?UGACUUCCACUGAAGGUAGUCCGUU?5′;siRNA3正义链:5′? GGUCUAUCCAUGACCUUCGUGUCAU?3′,反义链:3′?CCAGAUAGGUACUGGAAGCACAGUA?5′。转染操作的对照序列(按cDNA)为:CGCACGAACAACTCCCAGTCGACTT,同上设计出对照siRNA分子。本实验的siRNA均为25nt两端齐平设计。3个siRNA分子及其对照物均由Invitrogen公司化学合成,脱盐法提纯,5′?端行Cy3荧光基团标记。
1.2 其他主要试剂用品
Lipofectamine 2000(Invitrogen),DMEM培养液(Hyclone),兔抗大鼠GFAP抗体(Sigma),兔抗大鼠Vimentin抗体(博奥森),兔抗大鼠S100抗体(博奥森),兔抗大鼠CNTF抗体(博奥森),胰酶(Sigma),Cy3标记羊抗兔第二抗体(中杉金桥),反转录和PCR试剂盒 (Fermenta);DNA标准参照物(华美),24孔培养板(Costar)。
1.3 主要仪器
高压蒸汽灭菌器(Sanyo),倒置荧光显微镜(Zeiss),数码相机(Olympus,带专用订制接口)超净工作台(苏州净化),二氧化碳孵箱(Sanyo),PCR仪(Bio?Rad),水平电泳仪(北京六一),微量加样枪(DISHI),凝胶成像系统(ChemiDoc),低温冰箱 (Sanyo),离心机(Bio?Rad)。
1.4 实验动物发表论文网
成年健康SD大鼠,由四川大学实验动物中心提供。雌雄合笼使其自然交配。雌鼠孕期给予孕鼠全价饲料,饮水经煮沸消毒处理并可自由摄取,饲养间环境温度18~24 ℃,自然光照。分娩后3天(本实验3种细胞的培养均取自该日龄),取乳鼠行断颈处死,整体浸泡于70%乙醇30 min后,置于超净台中的无菌培养皿内,解剖取材进行细胞培养。
1.5 细胞培养
本实验分别培养了施万细胞、嗅鞘细胞和成纤维细胞。施万细胞取自坐骨神经干,操作程序参考Manson 等[6]的方法;待细胞形态完全恢复梭形(大约3 d)即进行传代,16 h后单独用Lipofectamine 2000模拟转染细胞(见“1.6”),继续培养72 h,用S100免疫组化鉴定并观察。嗅鞘细胞取自嗅球,采用李绍波等的操作程序[7],纯化后培养7 d进行传代,16 h后同上用Lipofectamine 2000处理,继续培养72 h,用GFAP与p75鉴定并观察。成纤维细胞取自肾脏,参考Alvarez等的方法[8],血清浓度5%,细胞融合率达90%时进行传代,传代后处理同上,用Vimentin免疫组化鉴定并观察。以上细胞在纯化培养完成后,消化转瓶的过程中不减少细胞总数;所有培养液体不加抗生素。细胞观察计数在倒置显微镜下拍照并用ImagePro 5.0软件辅助分析,记录每高倍(40×10)视野下细胞的面积占视野总面积的比例,作为细胞的融合度(conflurency);随机计数5个视野(视野不能含有培养板的底部边缘)后取平均值作为该孔的融合度;一共计数6个孔取平均作为本组的细胞融合度。
1.6 siRNA体外转染操作
成纤维细胞以3×105个/ml的密度传代一次后,待细胞融合率达50%时,常规胰酶消化,不分瓶直接重新接种于D?多聚赖氨酸铺底的24孔板,每孔加入不含血清的DMEM培养基0.2 ml。其中6个孔在传代后16 h时进入转染程序,另6个在传代后48 h时进行。转染时,所有液体、容器和有可能接触标本的用具都要用DEPC处理后行高压灭菌。首先,摇匀Lipofectamine 2000原液,取6 μl,用300 μl DMEM培养基稀释,摇匀后室温(20 ~ 25 ℃)孵育5 min。然后取20 μmol/L siRNA原液6 μl,用300 μl DMEM培养基稀释于Eppendorf管中,轻轻翻转混匀。将Lipofectamine 2000稀释液和siRNA稀释液混合,轻轻翻转混匀,室温下孵育20 min。在此期间如果液体变浑浊并不影响效果。弃去培养板孔中的培养基,立即加入上述混合液,每孔100 μl。对照siRNA的转染操作方法与上述相同。转染过程需要避强光。转染后,细胞置于原培养条件(37 ℃,5% CO2)下继续培养,6 h后在倒置荧光显微镜下观察是否成功转染;分24,48和72 h三个时间点取材检测,每个时间点检测12个孔,其中6个为待检siRNA孔,6个为对照孔。
1.7 RT?PCR分析
用Premier 5.0软件设计大鼠β?肌动蛋白和CNTF的引物序列。β?肌动蛋白引物上游序列:5′?GTAAAGACCTCTATGCCAACA?3′,下游序列:5′?GGACTCATCGTACTCCTGCT?3′,扩增产物预计227 bp;CNTF引物上游序列:5′?CTTTCGCAGAGCAAACACCT?3′,下游序列:5′?CATCCCATCAGCCTCATTTT?3′ ,扩增产物预计422 bp。按说明书指示的流程进行操作,反应体系的总体积为25 μl;PCR扩增条件:94℃预变性(3 min),以94℃变性45 s,51℃退火45 s,72℃延伸1 min为一个循环,35个循环后,72℃延伸5 min。15 g/L琼脂糖凝胶电泳分离扩增产物,在凝胶成像仪上观察拍照,用ImagePro 5.0软件测定并计算CNTF与β?肌动蛋白扩增产物条带的平均灰度与条带面积乘积的比值,作为CNTF mRNA的相对含量。发表论文网
1.8 免疫荧光组织化学检测
将细胞培养板内的液体倒掉,用D?Hanks液快速漂洗2次,加入4℃预冷的95%乙醇铺满培养板底面,固定15 min。倾去乙醇,用PBS漂洗3次,每次2 min。滴加2%牛血清白蛋白PBST溶液封闭15 min,倾去后不洗直接滴加用PBS 1∶200稀释的CNTF抗体工作液,37℃孵育1 h后转入4℃冰箱孵育16 h。PBS漂洗3次,每次5 min。滴加PBS 1∶200稀释的荧光二抗,37℃避光孵育1 h。PBS避光漂洗3次,每次3 min。倒置荧光显微镜下观察、测定细胞的平均荧光强度(灰度)值并照相。
1.9 数据统计分析
用SPSS 16.0软件进行数据分析,采用组间t检验,P<0.05认为差别具有统计学意义。数据导入Excel 2003绘制直方图。
2 结 果
2.1 经过传代和Lipofectamine 2000处理后成纤维细胞的融合度最高,符合转染要求
经过传代和Lipofectamine 2000处理,如图1所示,施万细胞的融合度大大下降,嗅鞘细胞的融合度也明显降低,两者都不能满足常规siRNA转染的要求。成纤维细胞经过两次传代,细胞密度增加,细胞生长良好,满足转染要求。
2.2 在传代后16 h时进行转染操作,siRNA能够成功进入成纤维细胞
转染操作6 h后观察,如图2,传代后16 h转染组成纤维细胞90%以上带有红色荧光标记,证实siRNA已经成功转染成纤维细胞。传代后48 h转染组成纤维细胞很少见到荧光标记。
2.3 不同siRNA分子和不同转染条件下CNTF免疫荧光强度的变化
如图3所示,在传代后16 h进行转染操作,转染后24,48,72 h这3个时间点,3种核苷酸序列不同的siRNA分子所致的CNTF免疫荧光强度降低的程度是不同的。其中,siRNA2组各时间点CNTF免疫荧光强度无显著改变;siRNA1和siRNA3组可见在72 h时CNTF免疫荧光强度下降显著(P<0.01),两组分别下降至对照siRNA转染组的82%和61%。
2.4 siRNA3转染后CNTF mRNA水平显著下降
如图4,5,经RT?PCR分析可见,siRNA3转染后24 h,成纤维细胞的CNTF mRNA已经显著降低到对照siRNA转染组的14%,此后一直维持低水平。
3 讨 论发表论文网
本实验的主要内容为RNA干扰技术中siRNA的设计和有效性检验环节的操作。通过一系列操作,成功筛选出针对大鼠CNTF的相对有效的 siRNA3分子,并在最佳条件下使体外培养的成纤维细胞的CNTF mRNA和蛋白水平分别降低到对照siRNA转染组的14%和61%。
在包括CNTF等多种分子在内的RNAi实验中,我们根据操作经验,总结出大致相似的如下几个步骤。首先,根据目标蛋白的mRNA全长序列,按照经验规则[3-5]选择候选靶序列及其对照序列,并完成同源性检测;靶序列一般选择 3个,如果后续试验发现全部无效再作补充,否则数量多了会大大增加开支。选择靶序列和设计siRNA有很多自动化工具可用,我们应用后认为 Invitrogen公司的BLOCK?iTTM RNAi Designer比较全面和方便。此外,像麻省理工学院Whitehead研究所的siRNA设计程序和日本基因网SiDirect siRNA序列分析程序等得出的结果也值得参考,以上程序都可以在互联网上免费使用。siRNA分子可以是传统的19nt加上?dTdT等突出尾巴,也可以不采用19nt突出结构,评判是否有效的唯一方法就是通过实验验证。第二,根据靶序列设计出siRNA分子,确定siRNA分子的修饰类型,并交由专门的公司或实验室合成;siRNA合成修饰不是本文的重点内容,大多数研究形态或者功能的实验室也不需要常规进行这些操作。第三,通过文献查阅确定哪些细胞表达目标蛋白,然后用体外培养预实验确定:这些细胞中哪种或哪些细胞既能表达目标蛋白又比较容易转染。需要注意的是,为了检测细胞的生长特性和存活能力,传代过程不要降低细胞的总数。比如本实验通过体外培养发现,成纤维细胞高表达CNTF,而且传代后经过Lipofectamine 2000作用,仍然能生长和增殖,最符合转染操作的要求。如果有现成的细胞株或细胞系可以利用,仍然有必要进行上述预实验,因为细胞系或细胞株在多次传代后有可能丢失某些分子特点,甚至于其来源和属性都可能存在疑问[9]。如果实验室已经有相应细胞的培养经验,这一步上将会节约大量时间。第四,细胞消化传代(植板时不要减少细胞密度)并于16 h左右进行转染操作,大约6 h后镜下观察。之所以要在消化后16 h左右进行转染,是因为经过实验我们发现,细胞生长时间过长或过短,常规脂质体转染的成功率会大大下降。转染后6 h首次观察的目的是借助siRNA分子修饰基团(如荧光基团或酶)的成像确定siRNA是否已经进入细胞内;成功转染的细胞数应该在细胞总数的70%以上才具有意义[10]。至于siRNA的有效浓度,我们认为有必要进行预实验。因为浓度太低没有明确的干扰效果,浓度太高会因Lipofectamine 2000用量的增加而致使较多的细胞死亡。本文采用的浓度就是CNTF体外干扰的最佳浓度(本文中未显示该项预实验的数据)。第五,在转染成功的前提下继续培养,分多个时间点对各组细胞进行目标蛋白的免疫荧光组织化学检测,选出干扰效果最好的siRNA分子。时间点的选择可从转染后24 h开始,到72 h结束,只要有效,一般都能看到荧光强度减弱。虽然在哺乳动物细胞,常规脂质体转染的RNAi由于siRNA不能在细胞内复制和长期存在[11],其维持时间往往不超过170 h,但在siRNA分子有效性检测的环节,时间点没有必要选择那么长。最后,设计目标蛋白对应mRNA的RT?PCR引物(如果能从文献上或本实验室的数据库中获得准确信息更好),用通过免疫荧光检测选出的最有效的siRNA进行体外RNAi操作,分时间点取出细胞进行RT?PCR检测,进一步验证 mRNA水平是否被显著降低。
我们在siRNA有效性验证实验中,并没有先检测mRNA再检测蛋白,而是先通过免疫荧光检测找出最能降低蛋白表达水平的siRNA分子。这样做的原因有两方面:其一,蛋白水平的变化直接与功能相关,后续实验中RNAi是否有意义和能否起到人工干预效果,取决于蛋白表达是否降低,而不只是 mRNA水平的下降;其二,RT?PCR不论从成本还是操作的繁琐程度来说,都要高于免疫荧光检测。因此,我们在有效性验证的常规实验中反其道而行之,先看蛋白变化并找出最有效的siRNA分子,再看这种变化是否有mRNA水平降低这个基础条件,从而最终肯定所选siRNA分子的干扰效能。这样既快捷又节省费用。如果看不到免疫荧光强度的明显改变,再做一下蛋白质印迹是值得的。某些分子的RNAi操作中,mRNA水平降低后,蛋白水平始终不下降。虽然有的体内实验尝试每隔24 h到48 h反复转染取得了效果[12],但是我们在实践中发现这一做法在体外实验中经常是不能见效的,Lipofectamine 2000可能有一定细胞毒性,多次转染后细胞数量有明显下降(本文未显示该数据),况且这样达到的干扰效果即使用到体内实验中也将没有实际用途。所以如果发现这类蛋白水平难以降低的目标蛋白分子,往往有必要考虑改变转染方法,甚至调整实验的技术路线。
siRNA的脂质体转染技术虽然比较“传统”,但目前是进行体外RNAi操作的最常规且相当简便的方法。除了用具与液体必须杜绝RNAse外并没有需要特别小心的问题。成功地验证一个功能性的siRNA分子,就意味着在某个目标蛋白的研究中多了一个工具,并且根据相应的靶mRNA序列还能够设计出同样有效的短发夹状RNA(shRNA)分子,通过病毒载体等其他手段进行RNAi实验。因此掌握了siRNA的设计和有效性验证方法,就为RNAi实验奠定了基础。但是,本文的方法并不适用于所有的分子。这套方法对于存在以下问题的分子不适合:①只在神经细胞或其他某些难以分离传代的细胞上表达,这种情况需要使用病毒载体进行转染;②mRNA序列不长缺少选择余地,或者选择的靶序列经实验验证无一有效,或者设计的siRNA与管家基因有冲突,这些情况解决起来尚有困难;③目标蛋白半衰期太长,或者细胞内有大量储备,一时难以消耗掉,这种情况下有可能观察不到免疫荧光强度的降低,如有必要需直接用 RT?PCR检测来筛选有功能的siRNA分子。
发表论文网
【参考文献】
[1] Dykxhoorn DM, Palliser D, Lieberman J. The silent treatment: siRNAs as small molecule drugs[J]. Gene Ther, 2006,13(6): 541-552.
[2] Behlke MA. Progress towards in vivo use of siRNAs[J]. Mol Ther, 2006, 34(4): 644-670.
[3] Aronin N. Target selectivity in mRNA silencing[J]. Gene Ther, 2006,13(6): 509-516.
[4] Schwarz DS, Hutvágner G, Du T, et al. Asymmetry in the assembly of the RNAi enzyme complex[J]. Cell, 2003, 115(2):199-208.
[5] Pei Y, Tuschl T. On the art of identifying effective and specific siRNAs[J]. Nature Methods, 2006, 3(9): 670-676.
[6] Mason PW, Atlema BL, De Vries GH. Isolation and characterization of neonatal Schwann cells from cryopreserved rat sciatic nerves[J]. J Neurosci Res, 1992, 31(4): 731-744.
[7] 李绍波, 章 为, 周 雪,等. 成年大鼠嗅鞘细胞的原代培养与鉴定[J]. 四川大学学报: 医学版, 2007, 38(5): 717-719.
[8] Alvarez RJ, Sun MJ, Haverty TP, et al. Biosynthetic and proliferative characteristics of tubulointerstitial fibroblasts probed with paracrine cytokines[J]. Kidney Int, 1992, 41(1): 14-23.
[9] 弗雷谢尼. 动物细胞培养: 基本技术指南[M]. 北京: 科学出版社, 2004: 438.
[10] Tuschl T, Zamore PD, Lehmann R, et al. Targeted mRNA degradation by double?stranded RNA in vitro[J]. Genes Dev, 1999, 13(24): 3191-3197.
[11] Zamore PD. RNA interference: listening to the sound of silence[J]. Nat Struct Biol, 2001, 8(9): 746-750.
发表论文网
[12] Dorn G, Patel S, Wotherspoon G, et al. siRNA relieves chronic neuropathic pain[J]. Nucleic Acids Research, 2004, 32(5): e49.